今天的工作做的不多,主要是後面又玩雲服務器去了,那個東西配置了小半天,華為的,學校花了大價錢搞過來的320G運行內存,結果因為是centos7,還是arch的 搞的我連anaconda的官方版都沒有,最後只能去GitHub搞一個適用arch的,Python版本只到3.7.1,然後我的代碼又是按照新語法來的,有類型,又不支持。
所以今天只做了基於Kmeans++ 來分種群,本來還想搞拓撲結構的,但是,那幾篇中文論文質量真的底下,也沒說清楚,我知道拓撲結構是咋樣的,但是用的是什麼策略獲取的相鄰的點的,看了半天啥也沒有,如果是基於距離的話,高緯度就不用做了。目前有可能的是按照粒子的ID來的。如果是那樣的話,我感覺不如直接亂來算了。
鄭重提示:本文版權歸本人所有,任何人不得抄襲,搬運,使用需征得本人同意!
日期:2022.6.21 DAY 2
昨天的時候編碼有個問題,一直沒發現,今天跑了幾次發現結果對不到,於是我拿來以前同學寫過的PSO對比了一下,一開始以為是np的問題,可能是哪裡深淺復制有問題,仔細看發現,深淺復制的話我使用的都是深拷貝(通過生成新的list實現),然後以為是np的問題,然後,我又按照我的代碼格式去寫(因為要上分布式,所以能不用矩陣就不用)後來發現其實是我的速度忘記更新給粒子了。
#coding=utf-8
#這個是最基礎的PSO算法實現,用於測試當前算法架構的合理性
#此外這個玩意還是用來優化的工具類,整個算法是為了求取最小值
import sys
import os
sys.path.append(os.path.abspath(os.path.dirname(os.getcwd())))
from ONEPSO.Bird import Bird
from ONEPSO.Config import *
from ONEPSO.Target import Target
import random
import time
class BasePso(object):
Population = None
Random = random.random
target = Target()
W = W
def __init__(self):
#為了方便,我們這邊直接先從1開始
self.Population = [Bird(ID) for ID in range(1,PopulationSize+1)]
def ComputeV(self,bird):
#這個方法是用來計算速度滴
NewV=[]
for i in range(DIM):
v = bird.V[i]*self.W + C1*self.Random()*(bird.PBestX[i]-bird.X[i])\
+C2*self.Random()*(bird.GBestX[i]-bird.X[i])
#這裡注意判斷是否超出了范圍
if(v>V_max):
v = V_max
elif(v<V_min):
v = V_min
NewV.append(v)
return NewV
def ComputeX(self,bird:Bird):
NewX = []
NewV = self.ComputeV(bird)
bird.V = NewV
for i in range(DIM):
x = bird.X[i]+NewV[i]
if(x>X_up):
x = X_up
elif(x<X_down):
x = X_down
NewX.append(x)
return NewX
def InitPopulation(self):
#初始化種群
GBestX = [0. for _ in range(DIM)]
Flag = float("inf")
for bird in self.Population:
bird.PBestX = bird.X
bird.Y = self.target.SquareSum(bird.X)
bird.PbestY = bird.Y
if(bird.Y<=Flag):
GBestX = bird.X
Flag = bird.Y
#便利了一遍我們得到了全局最優的種群
for bird in self.Population:
bird.GBestX = GBestX
bird.GBestY = Flag
def Running(self):
#這裡開始進入迭代運算
for iterate in range(1,IterationsNumber+1):
w = LinearW(iterate)
#這個算的GBestX其實始終是在算下一輪的最好的玩意
GBestX = [0. for _ in range(DIM)]
Flag = float("inf")
for bird in self.Population:
#更改為線性權重
self.W = w
x = self.ComputeX(bird)
y = self.target.SquareSum(x)
# 這裡還是要無條件更細的,不然這裡的話C1就失效了
# if(y<=bird.Y):
# bird.X = x
# bird.Y = y
bird.X = x
bird.Y = y
if(bird.Y<=bird.PbestY):
bird.PBestX=bird.X
bird.PbestY = bird.Y
#個體中的最優一定包含了全局經歷過的最優值
if(bird.PbestY<=Flag):
GBestX = bird.PBestX
Flag = bird.PbestY
for bird in self.Population:
bird.GBestX = GBestX
bird.GBestY=Flag
if __name__ == '__main__':
start = time.time()
basePso = BasePso()
basePso.InitPopulation()
basePso.Running()
end = time.time()
# for bird in basePso.Population:
# print(bird)
print(basePso.Population[0])
print("花費時長:",end-start)
這部分修改還是參考原來的那幾篇文獻來做的。
首先是Kmeans++的實現。
#coding=utf-8
#由於中心點的選取對於PSO多種群來說比較重要
#所以這裡選擇Kmeans++ 來慎重選擇中心點
#距離公式采用歐式距離
import sys
import os
sys.path.append(os.path.abspath(os.path.dirname(os.getcwd())))
from ONEPSO.Bird import Bird
from ONEPSO.Config import *
import random
import math
class KmeansAdd(object):
choice = random.sample
ClusterCenters = []
def Divide(self,Popluations,Centers):
#劃分,這邊也是如果不滿足條件就按照最後一個族群來劃分
#這裡注意一點原來的CID的編號是多少壓根就不重要,只要後面准確分類就沒有問題
for bird in Popluations:
BirdCID = -1
Flag = float("inf")
for center in Centers:
dist = self.EuclideanDistan(bird,center)
if(dist<=Flag):
Flag=dist
BirdCID+=1
bird.CID=BirdCID
bird.DIST=Flag
def EuclideanDistan(self,bird,center):
#歐式距離公式
x1 = bird.X
x2 = center
dist = 0
for index in range(len(x1)):
dist+=math.pow((x1[index]-x2[index]),2)
return dist
def InitChoiceCenter(self,Population):
#初始化選取中心點,先選擇一個,然後幫我完成初始化分類,此時的ClusterCenters存儲的就是
#我們初始化得到的中心點
K = 1
self.ClusterCenters = self.choice(Population,K)
RelCenters = []
for i in range(K):
#分配CID
self.ClusterCenters[i].CID=(i)
RelCenters.append(self.ClusterCenters[i].X)
#這個中心點存的是可行域內的點坐標
self.ClusterCenters=RelCenters
#此時會完成第一次劃分
self.Divide(Population,self.ClusterCenters)
#構造權重模型
Tim = ClusterNumber-K
for i in range(Tim):
K+=1
Weights = [bird.DIST for bird in Population]
Total = sum(Weights)
Weights=[weight/Total for weight in Weights]
#在采用輪盤算法
x = -1
i = 0
Due = random.random()
while(i<Due):
x+=1
i+=Weights[x]
self.ClusterCenters.append(Population[x].X)
#再次劃分中心點,之後通過增加中心點,我們可以完成對子種群的劃分
self.Divide(Population, self.ClusterCenters)
def RunningChoiceCenter(self,Population):
#運行時選取中心點,返回計算後的新的中心點
NewClusterCenterPoints={
}
Counter = {
}
#先初始化一下計數器,新的中心點
for CID in range(ClusterNumber):
Counter[CID]=0
for bird in Population:
if(not NewClusterCenterPoints.get(bird.CID)):
NewClusterCenterPoints[bird.CID]=bird.X
else:
NewClusterCenterPoints[bird.CID] = self.__ADD_X(NewClusterCenterPoints.get(bird.CID),bird.X)
Counter[bird.CID]=Counter[bird.CID] + 1
#計算出新的中心點
for CID in NewClusterCenterPoints.keys():
NewClusterCenterPoints[CID] = self.__Division_X(NewClusterCenterPoints.get(CID),Counter.get(CID))
return list(NewClusterCenterPoints.values())
def RunningDivide(self,Poplution,Iterate=0):
self.InitChoiceCenter(Poplution)
for epoch in range(K_Iterate):
NewClusterCenters = self.RunningChoiceCenter(Poplution)
if(
len(self.ClusterCenters)!=ClusterNumber or self.__SAME_Pred(self.ClusterCenters,NewClusterCenters)>=SAEMPRED
):
# print("Bad",epoch,Iterate,len(self.ClusterCenters))
break
else:
self.Divide(Poplution,NewClusterCenters)
self.ClusterCenters=NewClusterCenters
def __SAME_Pred(self,LastCenters,NewCenters):
#這個玩意是用來計算相似度的,如果發現相似度超過阈值,那麼停止迭代
res = 0.
count=0
if(len(LastCenters)!=len(NewCenters)):
return False
for i in range(len(LastCenters)):
for j in range(len(LastCenters[i])):
res+=math.pow((LastCenters[i][j]-NewCenters[i][j]),2)
count+=1
res = 1-(res/count)
return res
def __ADD_X(self,X1,X2):
res = []
for i in range(len(X1)):
res.append(X1[i]+X2[i])
return res
def __Division_X(self,X1,X2):
res =[]
for i in range(len(X1)):
res.append(X1[i]/X2)
return res
這個還是比較簡單的,主要是注意初始化和一般的Kmeans不一樣。然後這裡還是使用歐氏距離來做的,哪一個距離公式會好一點,這個要測試,我感覺是這個會好一點,因為,這個PSO本來就是基於空間的。
之後整合代碼如下:
#coding=utf-8
#現在是基於Kmeans++ 寫的動態劃分子種群的PSO算法。
import sys
import os
sys.path.append(os.path.abspath(os.path.dirname(os.getcwd())))
from ONEPSO.BasePso import BasePso
from ONEPSO.Config import *
from ONEPSO.Bird import Bird
import time
from ONEPSO.KmeansAdd import KmeansAdd
class DKMPSO(BasePso):
kmeansAdd = KmeansAdd()
def __init__(self):
super(DKMPSO,self).__init__()
self.kmeansAdd.RunningDivide(self.Population)
def ComputeV(self,bird):
#這個方法是用來計算速度滴
NewV=[]
for i in range(DIM):
v = bird.V[i]*self.W + C1*self.Random()*(bird.PBestX[i]-bird.X[i])\
+C2*self.Random()*(bird.GBestX[i]-bird.X[i]) + C3*self.Random()*(bird.CBestX[i]-bird.X[i])
#這裡注意判斷是否超出了范圍
if(v>V_max):
v = V_max
elif(v<V_min):
v = V_min
NewV.append(v)
return NewV
def ComputeX(self,bird):
NewX = []
NewV = self.ComputeV(bird)
bird.V = NewV
for i in range(DIM):
x = bird.X[i]+NewV[i]
if (x > X_up):
x = X_up
elif (x < X_down):
x = X_down
NewX.append(x)
return NewX
def InitPopulation(self):
#初始化種群
#這個是記錄全局最優解的
GBestX = [0. for _ in range(DIM)]
Flag = float("inf")
#還有一個是記錄Cluster最優解的
CBest = {
}
CFlag = {
}
for i in range(ClusterNumber):
CFlag[i]=float("inf")
for bird in self.Population:
bird.PBestX = bird.X
bird.Y = self.target.SquareSum(bird.X)
bird.PbestY = bird.Y
bird.CBestX = bird.X
bird.CBestY = bird.Y
if(bird.Y<=Flag):
GBestX = bird.X
Flag = bird.Y
if(bird.Y<=CFlag.get(bird.CID)):
CBest[bird.CID]=bird.X
CFlag[bird.CID] = bird.Y
#便利了一遍我們得到了全局最優的種群
for bird in self.Population:
bird.GBestX = GBestX
bird.GBestY = Flag
bird.CBestY=CFlag.get(bird.CID)
bird.CBestX=CBest.get(bird.CID)
def Running(self):
#這裡開始進入迭代運算
for iterate in range(1,IterationsNumber+1):
w = LinearW(iterate)
#這個算的GBestX其實始終是在算下一輪的最好的玩意
GBestX = [0. for _ in range(DIM)]
Flag = float("inf")
CBest = {
}
CFlag = {
}
for i in range(ClusterNumber):
CFlag[i] = float("inf")
for bird in self.Population:
#更改為線性權重
self.W = w
x = self.ComputeX(bird)
y = self.target.SquareSum(x)
# 這裡還是要無條件更細的,不然這裡的話C1就失效了
# if(y<=bird.Y):
# bird.X = x
# bird.PBestX = x
bird.X = x
bird.Y = y
if(bird.Y<=bird.PbestY):
bird.PBestX=bird.X
bird.PbestY = bird.Y
#個體中的最優一定包含了全局經歷過的最優值
if(bird.PbestY<=Flag):
GBestX = bird.PBestX
Flag = bird.PbestY
if (bird.Y <= CFlag.get(bird.CID)):
CBest[bird.CID] = bird.X
CFlag[bird.CID] = bird.Y
for bird in self.Population:
bird.GBestX = GBestX
bird.GBestY=Flag
bird.CBestY = CFlag.get(bird.CID)
bird.CBestX = CBest.get(bird.CID)
if((iterate+1)%100==0):
self.kmeansAdd.RunningDivide(self.Population,iterate)
if __name__ == '__main__':
start = time.time()
dkmpso = DKMPSO()
dkmpso.InitPopulation()
dkmpso.Running()
end = time.time()
# for bird in dkmpso.Population:
# print(bird)
print(dkmpso.Population[0])
print("花費時長:",end-start)
其實和原來的直接分沒太大區別,只是換了一個劃分的標准,然後這裡是每100次迭代重新劃分一次,其實我感覺就一開始,劃分後就不用劃分會好一點,這個也是測試會好一點。
首先,我們玩多種群的目的是,第一防止早熟,第二保證均勻性(後面上多目標,如果粒子都往一個方向跑,容前沿很難搞),第三 利用更多的信息,正對高緯度優化問題。
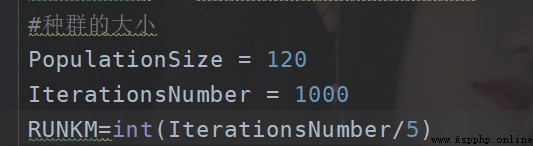
我這裡針對10個維度,然後大概對100,200,1000,2000,10000…次迭代做了實驗(大概20多個參數,然後每組大概10-20次實驗)。
然後設置每輪K次重新計算中心。
首先,總體上看,初始化劃分之後就不劃分了,效果是最差的。
之後是針對多少輪一次進行測試,大概目前得到的設置方式是,按照迭代次數除以5來是比較好的,也是比較快的。也就是五倍左右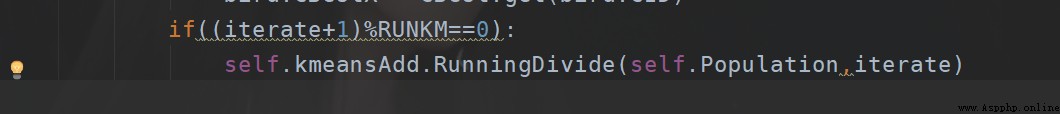
采用最傳統的粒子群的效果是最好的
采用我直接分子種群的效果不錯
采用這個有KMeans的效果和沒有的區別不大,差距不大,綜合情況下我推薦沒有的。不過這個目前的話Kmeans其實沒有用好,因為其實這個速度更新是和論文不一樣的。
當然這些都是在有限的實驗次數下,初步得到的結果,每個測試不跑個100+ 這個數據我說實話我是不太敢相信的。偶然性太高了。
之後是明天要研究這個拓撲結構的和強化學習的,那個拓撲結構的論文寫得是真模稜兩可,還有那篇介紹拓撲結構運用的,也是,不是看不起中文論文,是真的水分太大了。而且這中論文的學術造假可太容易做了,因為同樣的代碼,運行結果是不同的,稍微美化一下,數據就來了。
不過這裡的話,我其實還是在做各種測試,說實話,要不斷修改。
 [graduation project recommendation] Based on Django Dalian Housing source analysis and recommendation system
[graduation project recommendation] Based on Django Dalian Housing source analysis and recommendation system
【 Graduation project recommend
 Microsoft Visual c++ 14.0 or greater is required problem solution in Python
Microsoft Visual c++ 14.0 or greater is required problem solution in Python
Today I am writing a small pro