大家可以關注知乎或微信公眾號的share16,我們也會同步更新此文章。
疾病的預測往往能夠從病人的病歷歷史數據挖掘當中獲益,而許多有價值的發現也往往是在對於結構化數據的建模與分析之中得出的。通過結構化數據的預測與分析,判斷一名病人是否會出現急性肝功能衰竭的情況。
本次數據集,包含6000名20歲以上的成年人在2008-2009和2014-2015的兩次調查的情況。數據的采集來自’JPAC Center for Health Diagnosis and Control‘所做的,對全國范圍內的成年人的一次調查:通過專業人員的走訪研究,數據集涵蓋了范圍較廣的人口信息與他們的健康狀況信息,來源自直接訪談、身體檢查與血樣檢查(訓練集的比例為70%,測試集的比例為30%)。
點此下載數據集
本題是要預測病人是否會出現急性肝功能衰竭的情況,結果只有0-1兩種可能,所以我們可以采用K近鄰算法、樸素貝葉斯、邏輯回歸和決策樹這幾種算法。
補充:自動化參數調優,可以很大程度上減少工作量並提升工作效率。常用的兩種參數調優方法是暴力搜索和隨機搜索。
查重 * 缺失值處理 * 分類變量(如年齡分段處理等) * 將分類變量編碼為虛擬指標變量(pd.get_dummies)等,詳見文章’機器學習(一)‘的第2.1節內容。
import numpy as np
import pandas as pd
from pyecharts.charts import *
import pyecharts.options as opts
from pyecharts.commons.utils import JsCode
from sklearn.model_selection import train_test_split,cross_val_score
from sklearn.linear_model import LogisticRegression
from sklearn.tree import DecisionTreeClassifier
from sklearn.naive_bayes import BernoulliNB
from sklearn.metrics import roc_auc_score
X = pd.read_csv('/XXXXXX/訓練集.csv')
Y = pd.read_csv('/XXXXXX/測試集.csv')
''' 修改列名 '''
x_cols,y_cols = [],[]
for i in X.columns.to_list():
if '\u2028' in i:
x_cols.append(i.split('\u2028')[1])
elif '\n' in i:
x_cols.append(i.split('\n')[1])
else:
x_cols.append(i)
for i in Y.columns.to_list():
if '\u2028' in i:
y_cols.append(i.split('\u2028')[1])
elif '\n' in i:
y_cols.append(i.split('\n')[1])
else:
y_cols.append(i)
X.columns = x_cols
Y.columns = y_cols
''' 填充缺失值 '''
x_name = X.isna().sum()[X.isna().sum()>0].index.to_list()
y_name = Y.isna().sum()[Y.isna().sum()>0].index.to_list()
X[x_name] = X[x_name].fillna(X[x_name].mean())
Y[y_name] = Y[y_name].fillna(Y[y_name].mean())
''' 分類變量 '''
X['Age'] = pd.cut(X.年齡,bins=[19,30,41,52,63,74,85],\
labels=['(19,30]','(30,41]','(41,52]','(52,63]','(63,74]','(74,85]'])
Y['Age'] = pd.cut(Y.年齡,bins=[19,30,41,52,63,74,85],\
labels=['(19,30]','(30,41]','(41,52]','(52,63]','(63,74]','(74,85]'])
''' 劃分數據集 '''
x_new = pd.get_dummies(X,columns=['Age','性別','區域','護理來源']).drop('年齡',axis=1)
x_0 = x_new.drop(columns=['id','ALF'],axis=1)
y_0 = x_new[['id','ALF']]
x_train,x_test,y_train,y_test = train_test_split(x_0,y_0,test_size=0.3,random_state=2022)
''' 相關系數 '''
r = abs(x_new.corr().round(2).ALF).sort_values(ascending=False).drop('id')
r_name = list(r.index)
l = Line()
l.add_xaxis(list(r.index)[1:])
l.add_yaxis('',list(r)[1:],is_smooth=True,label_opts=opts.LabelOpts(is_show=False),
markpoint_opts=opts.MarkPointOpts(data=[opts.MarkPointItem(type_='max',name='最大值'),\
opts.MarkPointItem(type_='average',name='平均值')]))
l.set_global_opts(title_opts=opts.TitleOpts('ALF與各特征的相關系數',pos_top='10%',pos_left='center'))
l.render_notebook()
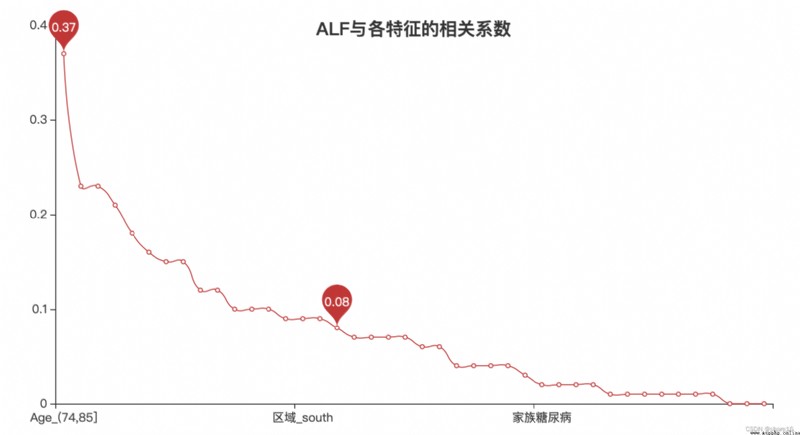
a = X[['id','Age','區域','性別','肝炎','高血壓','慢性疲勞','PVD','糖尿病','ALF']].round(2)
a_0 = a.groupby(['ALF','性別']).id.agg('count')
a_1 = a.groupby(['ALF','Age']).id.agg('count')
a_2 = a.groupby(['ALF','區域']).id.agg('count')
a_3 = a.groupby(['ALF','肝炎']).id.agg('count')
a_4 = a.groupby(['ALF','高血壓']).id.agg('count')
a_5 = a.groupby(['ALF','慢性疲勞']).id.agg('count')
a_6 = a.groupby(['ALF','PVD']).id.agg('count')
a_7 = a.groupby(['ALF','糖尿病']).id.agg('count')
a_11 = (a_1.loc[1,:]/a_1.reset_index().groupby('Age').id.agg('sum')*100).round(2).reset_index()
a_21 = (a_2.loc[1,:]/a_2.reset_index().groupby('區域').id.agg('sum')*100).round(2).reset_index()
a_31 = (a_3.loc[1,:]/a_3.reset_index().groupby('肝炎').id.agg('sum')*100).round(2).fillna(0)
a_41 = (a_4.loc[1,:]/a_4.reset_index().groupby('高血壓').id.agg('sum')*100).round(2).fillna(0)
a_51 = (a_5.loc[1,:]/a_5.reset_index().groupby('慢性疲勞').id.agg('sum')*100).round(2).fillna(0)
a_61 = (a_6.loc[1,:]/a_6.reset_index().groupby('PVD').id.agg('sum')*100).round(2).fillna(0)
a_71 = (a_7.loc[1,:]/a_7.reset_index().groupby('糖尿病').id.agg('sum')*100).round(2).fillna(0)
''' 性別/年齡/區域與ALF '''
p1 = Bar()
p1.add_xaxis(list(a_11.Age))
p1.add_yaxis('',list(a_11.id),bar_width='20%',label_opts=opts.LabelOpts(formatter='{c}%'),
color=JsCode("""new echarts.graphic.LinearGradient(0,0,0,1,\ [{offset:0,color:'#9DFEEC'},{offset:1,color:'#08B6D4'}],false)"""))
p1.set_global_opts(xaxis_opts=opts.AxisOpts(axisline_opts=opts.AxisLineOpts(is_show=False),
axistick_opts=opts.AxisTickOpts(is_show=False)),
yaxis_opts=opts.AxisOpts(is_show=False),
title_opts=opts.TitleOpts(title='患者年齡比例',subtitle='注:某年齡段患者人數/該年齡段的人數',
pos_top='1%',pos_right='10%'))
p2 = Bar()
p2.add_xaxis(list(a_21.區域))
p2.add_yaxis('',list(a_21.id),bar_width='20%',label_opts=opts.LabelOpts(formatter='{c}%'))
p2.reversal_axis()
p2.set_global_opts(xaxis_opts=opts.AxisOpts(is_show=False),yaxis_opts=opts.AxisOpts(is_inverse=True),
title_opts=opts.TitleOpts(title='患者區域比例',subtitle='注:某區域患者人數/該區域的人數',
pos_bottom='1%',pos_left='10%'))
p3 = Pie()
p3.add('',list((a_0.loc[1,:]/a_0.reset_index().groupby('性別').id.agg('sum')*100).round(2).to_dict().items()),
radius=['20%','30%'],center=['15%','35%'],
label_opts=opts.LabelOpts(position='inside',formatter='{b}:{c}%'))
p3.set_global_opts(title_opts=opts.TitleOpts(title='患者男女比例',subtitle='注:某性別患者人數/該性別的人數'),
legend_opts=opts.LegendOpts(pos_top='10%',pos_left='left'))
grid = Grid()
grid.add(p1,grid_opts=opts.GridOpts(pos_top='11%',pos_left='50%',pos_right='1%'))
grid.add(p2,grid_opts=opts.GridOpts(pos_top='50%',pos_bottom='10%',pos_left='5%'))
grid.add(p3,grid_opts=opts.GridOpts(pos_top='11%',pos_left='1%'))
grid.render_notebook()
性別/年齡/區域與ALF的圖表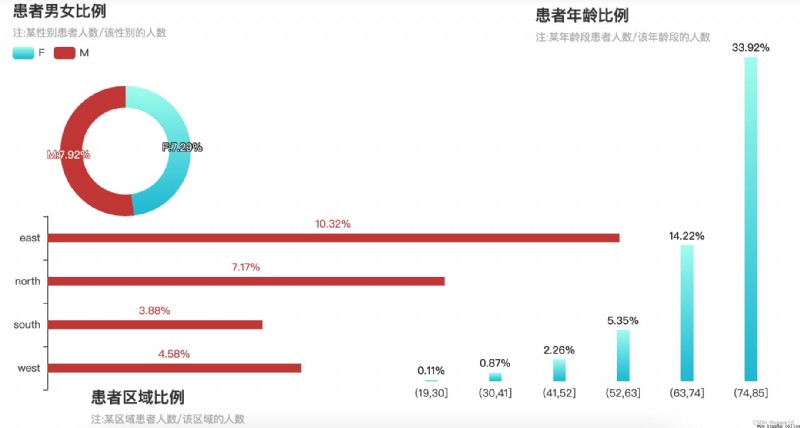

p4 = Pie()
p4.add('',list(a_31.to_dict().items()),radius=['10%','20%'],center=['15%','35%'],rosetype='area',
label_opts=opts.LabelOpts(formatter='{b}:{c}%'))
p4.set_colors(['#254EDB','#2CA127','#DBC72A'])
p4.set_global_opts(title_opts=opts.TitleOpts(title='ALF患者肝炎比例',subtitle='注:患者人數/總人數'),
legend_opts=opts.LegendOpts(pos_top='10%',pos_left='left'))
p5 = Pie()
p5.add('',list(a_41.to_dict().items()),radius=['10%','20%'],center=['45%','35%'],rosetype='area',
label_opts=opts.LabelOpts(formatter='{b}:{c}%'))
p5.set_colors(['#254EDB','#2CA127','#DBC72A'])
p5.set_global_opts(title_opts=opts.TitleOpts(title='ALF患者高血壓比例',pos_top='1%',pos_left='35%'),
legend_opts=opts.LegendOpts(pos_top='10%',pos_left='40%'))
p6 = Pie()
p6.add('',list(a_51.to_dict().items()),radius=['10%','20%'],center=['75%','35%'],rosetype='area',
label_opts=opts.LabelOpts(formatter='{b}:{c}%'))
p6.set_colors(['#254EDB','#2CA127','#DBC72A'])
p6.set_global_opts(title_opts=opts.TitleOpts(title='ALF患者慢性疲勞比例',pos_top='1%',pos_left='65%'),
legend_opts=opts.LegendOpts(pos_top='10%',pos_left='70%'))
p7 = Pie()
p7.add('',list(a_61.to_dict().items()),radius=['10%','20%'],center=['25%','85%'],rosetype='area',
label_opts=opts.LabelOpts(formatter='{b}:{c}%'))
p7.set_colors(['#254EDB','#2CA127','#DBC72A'])
p7.set_global_opts(title_opts=opts.TitleOpts(title='ALF患者PVD比例',pos_top='65%',pos_left='15%'),
legend_opts=opts.LegendOpts(is_show=False))
p8 = Pie()
p8.add('',list(a_71.to_dict().items()),radius=['10%','20%'],center=['60%','85%'],rosetype='area',
label_opts=opts.LabelOpts(formatter='{b}:{c}%'))
p8.set_colors(['#254EDB','#2CA127','#DBC72A'])
p8.set_global_opts(title_opts=opts.TitleOpts(title='ALF患者糖尿病比例',pos_top='65%',pos_left='45%'),
legend_opts=opts.LegendOpts(is_show=False))
grid = Grid()
grid.add(p4,grid_opts=opts.GridOpts(pos_top='20%',pos_left='10%'))
grid.add(p5,grid_opts=opts.GridOpts(pos_top='20%',pos_left='10%'))
grid.add(p6,grid_opts=opts.GridOpts(pos_top='20%',pos_left='10%'))
grid.add(p7,grid_opts=opts.GridOpts(pos_top='20%',pos_left='10%'))
grid.add(p8,grid_opts=opts.GridOpts(pos_top='20%',pos_left='10%'))
grid.render_notebook()
肝炎/高血壓等與ALF的圖表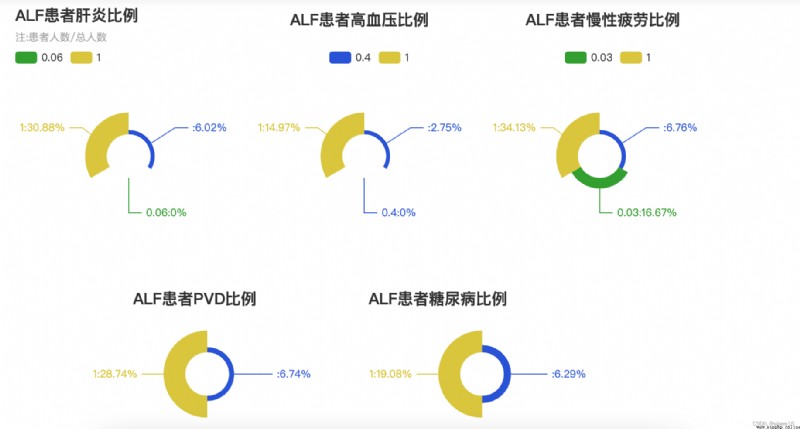
''' 交叉驗證,選出K個特征 '''
score = []
lr = LogisticRegression(solver='lbfgs',max_iter=6000)
for i in range(2,45):
s = cross_val_score(lr,x_train.loc[:,r_name[1:i]],y_train.iloc[:,1]).mean()
score.append(s)
k = range(2,45)[score.index(max(score))]
''' 暴力搜索-KNN '''
parameter = {
'n_neighbors':range(2,22)}
clf2 = GridSearchCV(KNeighborsClassifier(),parameter,cv=10)
clf2.fit(x_train.loc[:,r_name[1:k]],y_train.iloc[:,1])
print('暴力搜索的最優參數組合:{}'.format(clf2.best_params_))
print('暴力搜索的最優學習器評分:',clf2.best_score_)
y_new = pd.get_dummies(Y,columns=['Age','性別','區域','護理來源']).drop('id',axis=1)
df = pd.DataFrame(data=clf2.predict(y_new.loc[:,r_name[1:k]]),index=Y.id,columns=['ALF']).reset_index()
#df.to_csv('/XXXXXX/df.csv',index=False)
#print('打印完成!')
df.ALF.sum()
運行結果:
暴力搜索的最優參數組合:{‘n_neighbors’: 11}
暴力搜索的最優學習器評分:0.9231292517006804
預測的患者人數:5
''' 貝葉斯-二項分布 '''
clf1 = BernoulliNB()
clf1.fit(x_train.loc[:,r_name[1:k]],y_train.iloc[:,1])
print('貝葉斯得分:',clf1.score(x_train.loc[:,r_name[1:k]],y_train.iloc[:,1]))
y_pre = clf1.predict(x_test.loc[:,r_name[1:k]])
print('貝葉斯auc值:',roc_auc_score(y_test.iloc[:,1],y_pre))
''' 預測新數據 '''
y_new = pd.get_dummies(Y,columns=['Age','性別','區域','護理來源']).drop('id',axis=1)
df = pd.DataFrame(data=clf1.predict(y_new.loc[:,r_name[1:k]]),index=Y.id,columns=['ALF']).reset_index()
#df.to_csv('/XXXXXX/df.csv',index=False)
#print('打印完成!')
df.ALF.sum()
運行結果:
貝葉斯得分:0.8826530612244898
貝葉斯auc值:0.7482117440969309
預測的患者人數:201
''' 邏輯回歸 '''
lr.fit(x_train.loc[:,r_name[1:k]],y_train.iloc[:,1])
print('邏輯回歸得分:',lr.score(x_train.loc[:,r_name[1:k]],y_train.iloc[:,1]))
y_pre = lr.predict(x_test.loc[:,r_name[1:k]])
print('邏輯回歸auc值:',roc_auc_score(y_test.iloc[:,1],y_pre))
''' 預測新數據 '''
y_new = pd.get_dummies(Y,columns=['Age','性別','區域','護理來源']).drop('id',axis=1)
df = pd.DataFrame(data=lr.predict(y_new.loc[:,r_name[1:k]]),index=Y.id,columns=['ALF']).reset_index()
#df.to_csv('/XXXXXX/df.csv',index=False)
#print('打印完成!')
df.ALF.sum()
運行結果:
邏輯回歸得分:0.9258503401360544
邏輯回歸auc值:0.5595416225685194
預測的患者人數:35
''' 決策樹 '''
clf = DecisionTreeClassifier()
clf.fit(x_train.loc[:,r_name[1:k]],y_train.iloc[:,1])
print('決策樹得分:',clf.score(x_train.loc[:,r_name[1:k]],y_train.iloc[:,1]))
y_pre = clf.predict(x_test.loc[:,r_name[1:k]])
print('決策樹auc值:',roc_auc_score(y_test.iloc[:,1],y_pre))
''' 預測新數據 '''
y_new = pd.get_dummies(Y,columns=['Age','性別','區域','護理來源']).drop('id',axis=1)
df = pd.DataFrame(data=clf.predict(y_new.loc[:,r_name[1:k]]),index=Y.id,columns=['ALF']).reset_index()
#df.to_csv('/XXXXXX/df.csv',index=False)
#print('打印完成!')
df.ALF.sum()
運行結果:
決策樹得分:1.0
決策樹auc值:0.6259534323564834
預測的患者人數:141
經上述可知,
① 女性患者略高於男性;
② 年齡越大,出現急性肝功能衰竭狀況的可能性越高,年紀在74~85歲之間,有疾的概率可達34%;
③ 當人有肝炎、慢性疲勞、PVD的狀況時,有疾的概率可達30%;
④ 此次預測中,樸素貝葉斯-二項分布的auc值最好;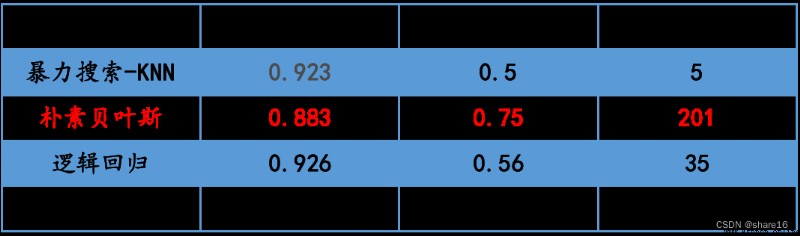
謝謝大家